
微生物总是生活在不断变化的环境中。为了生存,细菌建立了适应这些变化的能力。其中,代谢通量的重排和蛋白质组的重分配是细菌应对环境变化的常用策略[1-2]。比如,当外部营养条件改变时,细胞会及时调整内部代谢活动以合成氨基酸前体等关键代谢物,进而引起蛋白质组中不同种类蛋白质(例如转运蛋白、核糖体蛋白和代谢酶)的合成速率。我们知道,细胞内的化学反应绝大多数属于酶促反应。因此,蛋白质组中各蛋白组分的重分配反过来又会影响代谢网络中通量的分布。然而,现有的工作对细胞代谢通量和蛋白质分配的研究通常是单独开展的。因此,我们对细胞如何通过交叉调控其代谢通量和蛋白质合成以适应不断变化的营养条件这一科学问题还缺乏充分的理解。

文章上线截图
文章链接:https://doi.org/10.1016/j.ymben.2024.01.008
北京时间2月2日,中国科学院深圳先进技术研究院合成生物学研究所傅雄飞团队在Metabolic Engineering上发表题为“Cross-regulation between proteome reallocation and metabolic flux redistribution governs bacterial growth transition kinetics”研究成果。研究团队开发了一种名为受蛋白质分配约束的动态流平衡分析新方法(dCAFBA)(图1)。该方法利用代谢流平衡方法[3]描绘代谢状态的改变,借助粗粒化模型[4]模拟蛋白质资源分配的变化,并将酶对代谢反应速率的约束与中心底物代谢速率对蛋白质分配的调节相结合,建立了一个全面的模型,用于预测细菌对外界环境变化的响应。这种方法的独特之处在于,即便没有详尽的酶参数信息,也能成功预测大肠杆菌在面临营养条件变化时代谢通量重分布的动态变化过程。因此,该模型具有广泛的应用潜力。
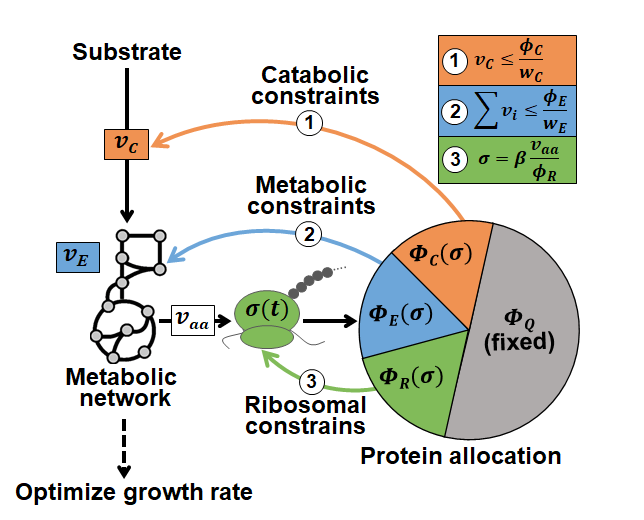
图1. dCAFBA方法
深入研究中,科研人员将dCAFBA模型预测的代谢通量动力学数据与已发表的蛋白质组数据进行比对,从而揭示了细菌是如何调整新陈代谢和蛋白质合成以适应不同营养条件变化的。研究发现,在营养丰富时期,计算得到的代谢通量变化与实验观测到的蛋白质水平动态变化相吻合;而在营养匮乏阶段,则揭示了一种由碳摄取蛋白向代谢酶转化的代谢瓶颈切换现象。这种切换破坏了代谢通量与蛋白表达水平间的协调关系,导致了先前研究未曾关注的增长过冲效应。
此外,研究团队将dCAFBA方法进行了扩展,研究了有价值化合物的异源合成对细胞生长动力学的影响。他们以番茄红素(lycopene)为例[5],模拟预测了在大肠杆菌中外源诱导表达番茄红素合成基因后细胞生长动力学的变化。该成果为设计实时调节关键酶表达的遗传回路以及提高代谢产物产量提供了重要的模型工具。
该研究揭示了微生物如何精巧地调控其内部代谢网络和蛋白质合成这一耦合系统,以适应不断变化的环境条件。研究中开发的dCAFBA方法不仅有助于深化我们对微生物生理机制的理解,也为研究代谢途径的动态调控提供了理论工具。
中国科学院深圳先进院合成所研究员傅雄飞和副研究员白阳为该文章的共同通讯作者,助理研究员袁慧丽和副研究员白阳为该文章的共同第一作者,合成所副研究员李雪飞在模型分析工作中做出了重要贡献。该研究得到国家重点研发计划、国家自然科学基金、中科院先导B类专项、广东省基础与应用基础研究基金自然科学基金、深圳合成生物学创新研究院及深圳合成生物研究重大科技基础设施等多个项目的支持。
PI简介
傅雄飞,研究员,博士生导师,中国科学院深圳先进技术研究院合成生物学研究所所长,定量合成生物学重点实验室(中国科学院)副主任,深圳合成生物学创新研究院副院长。
课题组主要研究方向:通过定量理论与合成重构结合,围绕胞内基因回路与底盘相互作用、空间扩张生物系统的有序结构设计原理等问题开展研究。
课题组现招聘:具有数学、物理、生物学、复杂系统等相关研究背景博士后、研究助理、技术员、客座学生等,欢迎感兴趣的朋友联系yang.bai@siat.ac.cn,简历及邮件标题注明“应聘岗位-学校名称-专业-姓名”。
实验室主页:
http://isynbio.siat.ac.cn/fulab
参考文献:
1. Jens Nilsen (2003) It is all about metabolic fluxes. Journal of Bacteriology 185, 7031-7035.
2. You CH, et al. (2013) Coordination of bacterial proteome with metabolism by cyclic AMP
signalling. Nature 500(7462):301-306.
3. Orth, J. D., Thiele, I. & Palsson, B. O. What is flux balance analysis? Nature Biotechnology 28,
245-248 (2010).
4. Erickson DW, et al. (2017) A global resource allocation strategy governs growth transition
kinetics of Escherichia coli. Nature 551(7678):119-+.\
5. Choi, H.S., et al., <i>In Silico</i> Identification of Gene Amplification Targets for Improvement of Lycopene Production. Applied and Environmental Microbiology, 2010. 76(10): p. 3097-3105.