
基因组规模代谢模型(Genome-scale metabolic model,GEM)是系统描述细胞代谢的数学模型,通过计算机建模开展“干”实验可以指导和部分代替费时费力或无法实现的“湿”实验,大幅提高研究效率。迄今为止,研究人员已经构建了数以千计物种的GEM,这些模型被广泛应用于生物制造和生命健康等领域,特别是在细胞工厂设计和人类疾病研究等方面发挥重要作用。
当前GEM的短板在于缺乏细胞生理信息约束,因此研究人员长期致力于在GEM中整合各类信息以改善模型。其中,整合酶信息的模型,即酶约束模型(Enzyme-constrained model,ecModel),相比于经典GEM,具有更好的预测性能和更广阔的应用前景。然而,一方面整合酶约束需要大量酶参数,而参数匮乏问题阻碍了酶约束模型的普及;另一方面,酶约束模型的构建和使用存在较高门槛,不利于模型的推广应用。
北京时间2024年1月18日,中国科学院深圳先进技术研究院陈禹与查尔姆斯理工大学Eduard J. Kerkhoven等人合作在Nature Protocols发表文章“Reconstruction, simulation and analysis of enzyme-constrained metabolic models using GECKO Toolbox 3.0”。该文章发布了酶约束模型构建和使用的最新版工具箱GECKO 3.0,优化了旧版[1, 2]并加入了酶参数预测[3]和选择模块,使得构建任意物种的酶约束模型成为了可能。另外,该文章作为GECKO 3.0的使用指南,详细描述了酶约束模型构建、模拟和分析的全部流程,能够大幅降低酶约束模型构建和使用门槛。文章第一作者是陈禹、Johan Gustafsson和Albert Tafur Rangel,通讯作者是Eduard J. Kerkhoven。

文章上线截图
文章链接:https://www.nature.com/articles/s41596-023-00931-7
首先,文章介绍了酶约束模型的原理(图1)以及GECKO 3.0的新功能,如增加了酶参数预测和选择的模块以及构建轻便版酶约束模型(light ecModel)的选项。
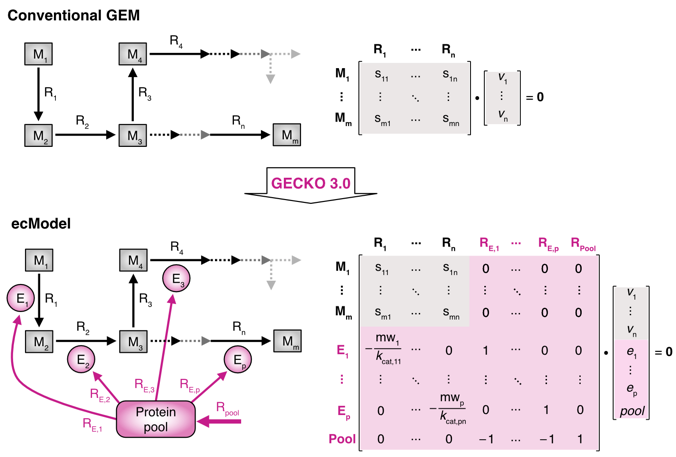
图1. 酶约束模型原理
随后,文章提供了使用GECKO 3.0的详细教程,包括文件和数据准备阶段(Stage 0),酶约束模型构建阶段(Stages 1–4,见图2)以及模型模拟分析阶段(Stage 5),总共涉及79个步骤。此外,文章以酿酒酵母Saccharomyces cerevisiae和人Homo sapiens酶约束模型的构建和使用为例对每一步进行了细致讲解。
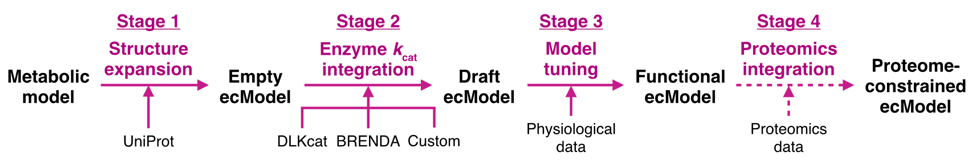
图2. 酶约束模型构建流程
最后,文章的预期结果(Anticipated results)和故障排除(Troubleshooting)部分能够帮助用户进行自查和调试。此外,伴随文章发布的代码仓库(https://github.com/SysBioChalmers/GECKO/)提供了在线交流平台,保障了用户和开发者的高效沟通。
总而言之,该文章为酶约束模型的构建和使用提供了便利,能够促进下一代代谢模型的普及。
PI与课题组简介
陈禹,研究员,博士生导师,入选国家重大人才工程青年项目(海外)。2018年获得华东理工大学生物化工博士学位,后于瑞典Jens Nielsen课题组开展博士后研究,2023年加入中国科学院深圳先进技术研究院合成生物学研究所。近五年一作文章发表于PNAS(4篇)、Nature Protocols、Molecular Systems Biology,Nucleic Acids Research等期刊,合作文章发表于Cell、Nature Catalysis、Nature Chemical Biology等期刊。多次受邀担任PNAS、Nature Communications等期刊审稿人。目前主持深圳市医学研究专项,参与国家重点研发计划、深圳市科技计划等项目。
陈禹课题组(https://chenyu.website/)从事系统生物学和合成生物学研究,聚焦基于代谢网络的数字细胞建模,整合“干”“湿”实验技术,实现细胞精准模拟以指导理性改造和人工合成;现招聘有生物工程、生物化工、发酵工程、生物信息学、计算生物学等相关研究背景博士后和研究助理各2-3名,欢迎有志之士投送简历至y.chen3@siat.ac.cn。
参考文献
[1] Sánchez BJ, et al. Molecular Systems Biology, 2017, 13(8): 935.
[2] Domenzain I, et al. Nature Communications, 2022, 13(1): 3766.
[3] Li F, et al. Nature Catalysis, 2022, 5(8): 662-672.