
4月17日,中国科学院深圳先进技术研究院司同课题组在国际学术期刊ACS Synthetic Biology发表研究论文“Deep Mutational Scanning of an Oxygen-Independent Fluorescent Protein CreiLOV for Comprehensive Profiling of Mutational and Epistatic Effects”。非氧依赖荧光蛋白CreiLOV是研究厌氧生物体系的重要工具。该研究团队依托深圳合成生物研究重大科技基础设施,基于FACS-seq方法表征其序列-功能关系,构建了包含15个位点20个突变的组合饱和突变文库(理论库容量18.4万)。应用机器学习模型,研究团队实现了基于少量低阶(两-两,三-三)突变数据预测15个位点间的高阶组合突变效应;在最优情况下,仅需覆盖理论设计空间0.25%的实验数据,实现全部组合突变空间的可靠预测。本研究为机器学习辅助的蛋白质工程实践提供新颖工具、经验数据和理论指导。课题组助理研究员陈永灿博士为本文的第一作者,司同研究员和张建志助理研究员为本文通讯作者。本文被收录在期刊主编Huimin Zhao教授组织的AI for Synthetic Biology特别专刊 (Virtual Special Issue)。

文章上线截图
文章链接:https://pubs.acs.org/doi/10.1021/acssynbio.2c00662
传统绿色荧光蛋白GFP发色团的成熟过程依赖氧气,无法应用于肠道微生物、肿瘤内部、无氧发酵等生物体系的研究。与GFP不同,黄素单核苷酸荧光蛋白(Flavin mononucleotide (FMN)-based fluorescent protein, FbFP)发色团成熟不需要氧气参与,在研究厌氧生物过程方面具有重大潜力。FbFP源于光敏蛋白的光-氧-电压结构域(light-oxygen-voltage domain, LOV domain)。天然LOV结构域在受到蓝光或紫外光激发时,FMN与其结合口袋内一个保守的半胱氨酸残基形成共价加合物,伴随着荧光消失和构象变化;暗环境下共价加合物衰变,荧光恢复。当该半胱氨酸突变为丙氨酸后,可将LOV结构域改造为稳定的FbFP,最大荧光发射波长为495 nm。FbFP具有分子量低、单体性、发色团成熟速度快、pH和热稳定性高等优点。然而,FbFP荧光强度和量子产率与GFP相比偏低,需要进行蛋白质工程。之前针对FbFP的工程改造多采用易错PCR、定点突变等传统定向进化方法,序列空间探索程度有限。
深度突变扫描(deep mutational scanning, DMS)通过集成大规模突变文库构建、高通量筛选和NGS测序,可对蛋白突变体序列-功能关系进行系统分析。目前,大量蛋白工程研究都利用单点饱和突变扫描文库进行深度突变扫描,大大提高了覆盖的突变位点和类型。然而,优良蛋白性能常需引入多个氨基酸突变。突变之间可能存在上位效应(epistasis),即两个或多个突变的效应不同于各自效应的总和。因此,即使已知所有单点突变效应,多点突变的理性设计也具有挑战性。优势突变组合虽然可以通过多轮定向进化积累,然而由于突变之间可能存在符号或双向符号上位效应,该贪心策略可能陷入局部最优。
在本研究中,作者以来源于莱茵衣藻的CreiLOV为研究对象,采用NNK简并密码子构建了118个位点的单位点饱和突变文库(理论库容2360)。为了获得序列-荧光强度数据,利用荧光激活细胞分选测序(FACS-seq)及表型估计方法快速表征,通过过滤的2185个突变序列占理论库容的92%以上。作者还对多种表型估计方法进行了比较,结果表明采用简单加权平均法各生物学重复相关性及估计与测量值相关性最高,其次为基于Gamma分布和正态分布的最大似然估计。根据序列-荧光强度数据,作者鉴定了减弱或增强CreiLOV荧光强度的关键位点、区域和氨基酸突变(图1)。
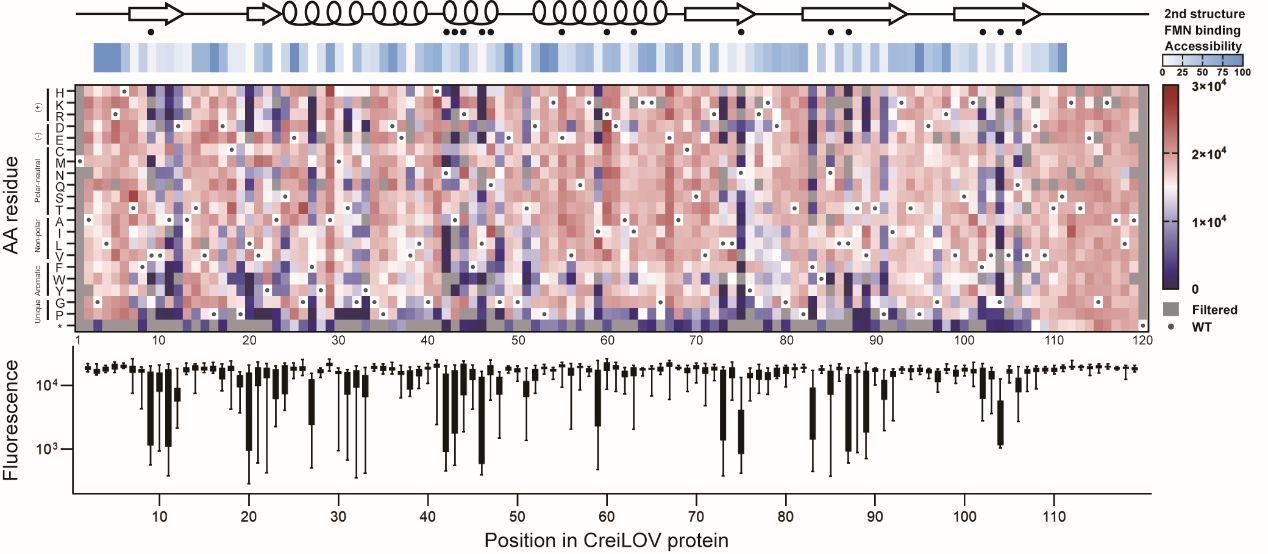
图1 CreiLOV单点突变效应分析
基于单点饱和突变扫描结果,作者进一步构建了覆盖15个位点的20个氨基酸突变的饱和突变组合文库,理论库容为18.4万。利用更大规模的FACS-seq,作者对多点突变体序列-荧光强度关系进行了解析,通过过滤的16.5万条突变序列约占理论库容的90%。作者发现,整体荧光强度随着突变位点数的增加而逐渐减弱。由于各突变均具有荧光增强或中性效应,表明存在广泛的负上位效应。对氨基酸突变之间特异性上位效应(specific epistasis)的统计分析验证了这一推论(图2)。
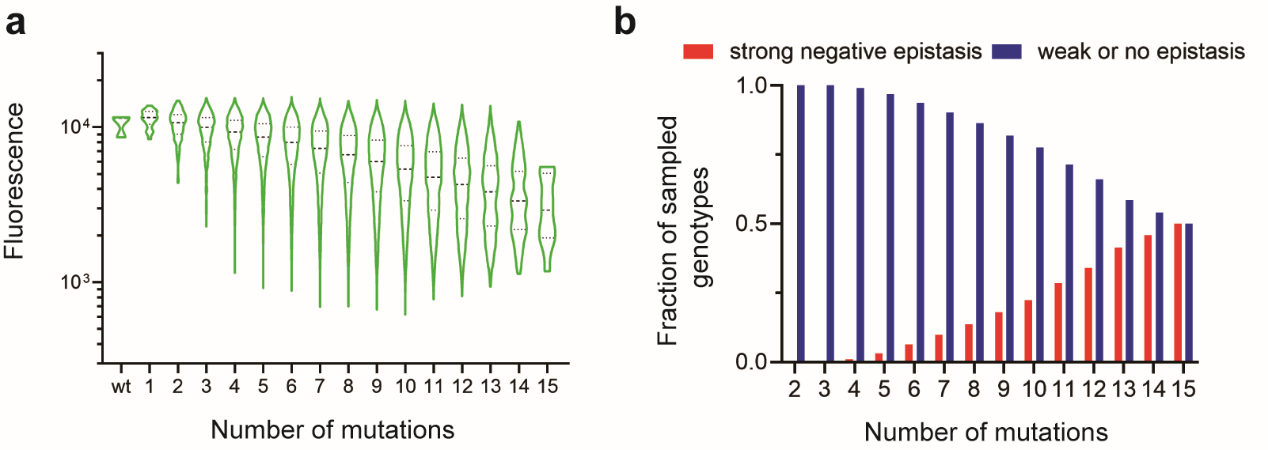
图2 CreiLOV(a)组合突变体表型分布与(b)特异性上位效应分析
近年来,科学家们发现对突变效应的解释还受到非特异性上位效应(nonspecific epistasis,也称为全局上位效应(global epistasis))的影响。非特异性上位效应是由于物理性质与生物效应之间存在非线性关系,是基因型-表型图(genotype-phenotype map, G-P map)的普遍特征。忽略该非线性关系常造成特异性上位效应的过度估计。MAVE-NN是最近开发的一种定量建模策略,整合了基因型-表型图模型、全局上位效应模型和噪声模型,并利用来源于信息论的三种互信息指标对模型性能进行度量。作者利用MAVE-NN对组合饱和突变数据集进行了定量建模,发现无论是基于基因型-表型图的加性模型还是黑箱模型,CreiLOV潜在表型与测量结果之间均呈现S型关系。考虑到该非线性特征,模型预测值与实验测量值具有高度相关性(图3)。
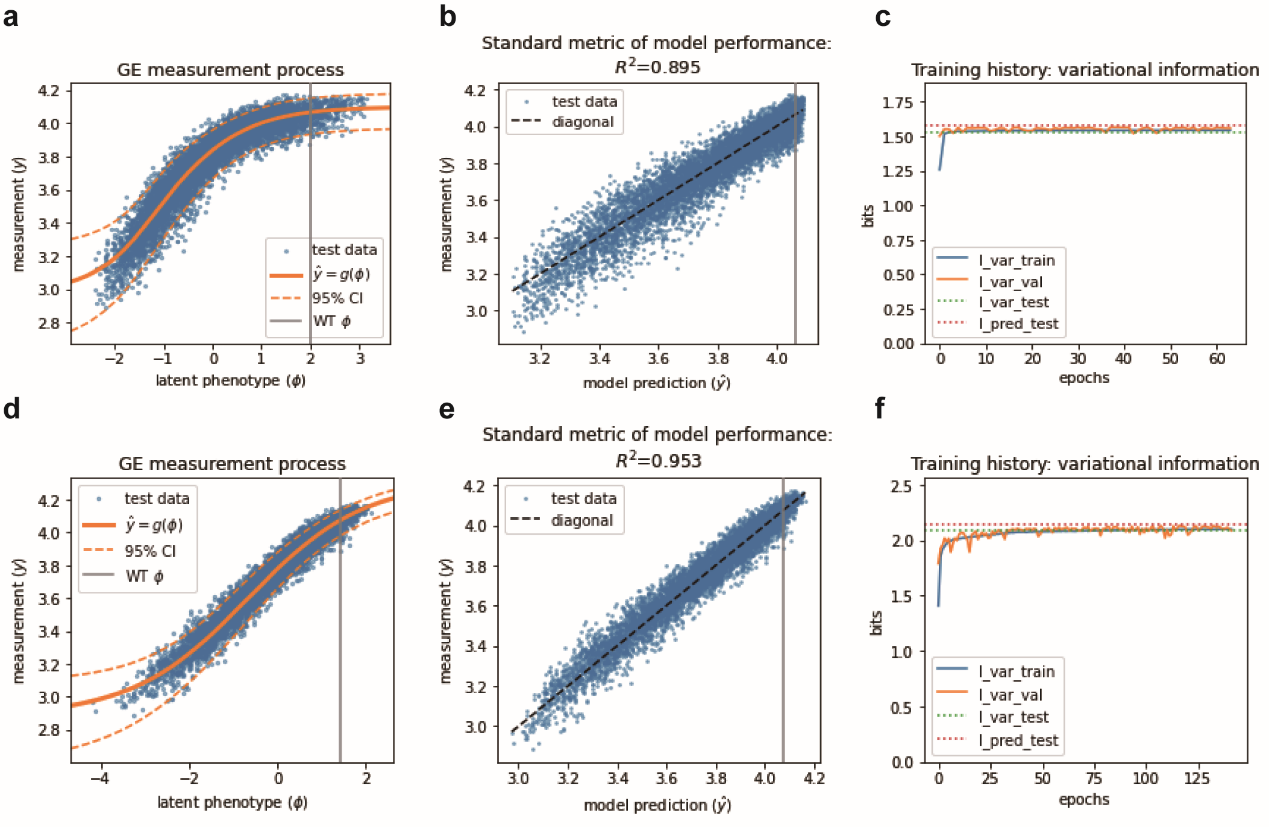
图3 CreiLOV非特异性上位效应建模与表型预测:(a-c)基于G-P图加性模型;(d-f)基于G-P图黑箱模型
如前所述,为获得更优的表型常需要在氨基酸序列中引入多点组合突变,而组合爆炸(combinatorial explosion)问题将为理性设计和实验测试均带来极大挑战。为了探究能否利用低阶突变体数据集预测高阶突变组合效应,作者使用1/2/3/4/5阶突变体数据对MAVE-NN机器学习模型进行训练,发现使用3阶及以下突变体数据集预测6阶以上突变效应时,模型预测与实验结果的Pearson相关系数可达0.84。值得注意的是,在仅使用1-3阶突变体数据集中10%的子集时,即可实现相对准确的预测(Pearson相关系数0.79)(图4)。进一步,作者利用文献报道的其他机器学习模型ECNet和其他组合突变数据集(CR9114和avGFP),探究了利用低阶突变数据预测高阶突变组合效应的普适性和限制因素。

图4 CreiLOV高阶突变体表型预测:(a)1/2/3/4/5及以下低阶突变体数据集预测6阶及以上突变体表型;(b)不同比例的3阶及以下突变体数据预测6阶及以上突变体表型
最后,作者还对单点和组合饱和突变文库进行多轮FACS筛选,获得了多个单点和多点突变体,其体内荧光强度和体外荧光量子产率显著提高(最高荧光量子产率达0.57),在60℃下热稳定性亦得到提高(图5),具有潜在应用价值。
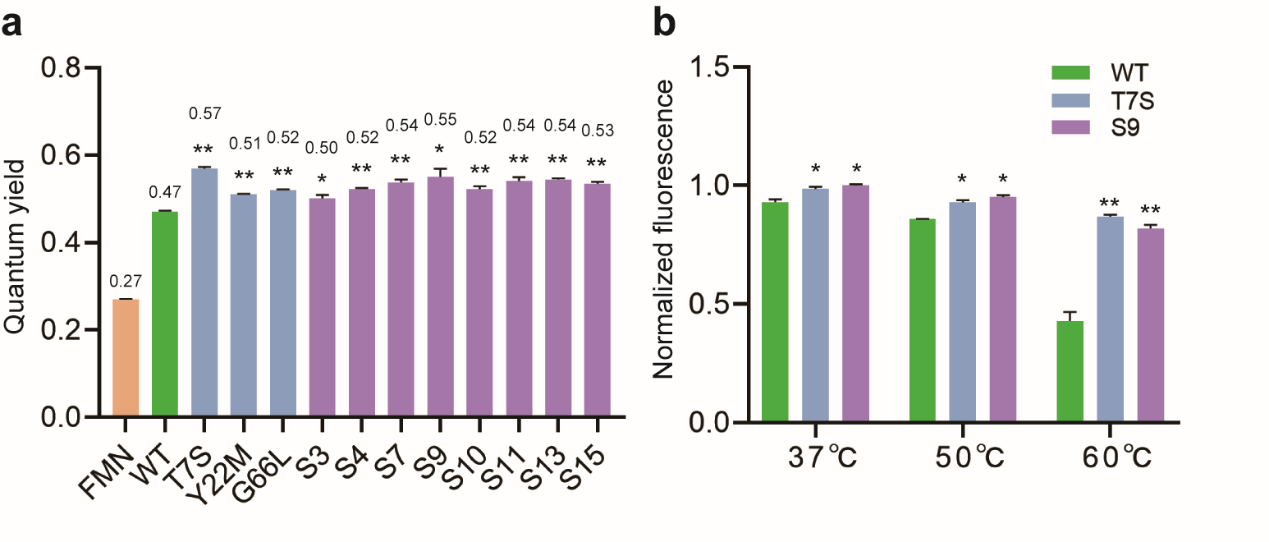
图5 CreiLOV优势突变体表征:(a)荧光量子产率;(b)热稳定性
综上,文章针对单位点和多位点组合饱和突变文库开展深度突变扫描,描绘了CreiLOV氨基酸突变效应和上位效应,筛选到得到性能显著提升的CreiLOV突变体。此外,作者还展示了机器学习模型基于少量低阶突变数据预测高阶突变体表型的可行性,为机器学习辅助的蛋白质工程优化设计提供重要的参考和指导。
该成果得到国家重点研发计划(2020YFA090023和2021YFA0910800)、国家自然科学基金(32071428)、广东省基础与应用基础研究基金(2021A1515110722)及深圳合成生物学创新研究院的支持。作者特别致谢中国科学院深圳先进技术研究院戴磊研究员关于DMS数据分析的讨论,以及清华大学张翀教授在FACS方面的帮助。